Bausteine von Vererbung und Verhalten: Gene, DNA, RNA, Proteine und Co
Wir stellen hier in sehr komprimierter Weise die Elemente der genetischen Informationsübertragung dar. Dieser Beitrag basiert auf verschiedenen – wesentlich ausführlicheren – Artikeln der deutschen Wikipedia und The Sequence Onotolgy.
Zu den neurologischen Grundlagen (Neuronen, Gliazellen, Synapsen, Neurotransmitter, Hormone, Rezeptoren, Aktionspotential, Blut-Hirn-Schranke siehe unter Neurologische Grundlagen
- 1. Gen
- 2. DNA (Desoxyribonukleinsäure)
- 3. RNA (Ribonukleinsäure)
- 4. Nukleotide
- 5. Proteine (Eiweiße)
- 6. Aminosäuren (Aminocarbonsäuren)
1. Gen
Ein Gen ist ein Abschnitt auf der DNA.
Die Gene in der DNA enthalten die Information zur Herstellung der RNA (Ribonukleinsäuren).
Protein-codierende Gene codieren mRNA (messenger RNA). Die mRNA enthält die Information zum Bau von Proteinen.
Die Abfolge der Basen bestimmt die Abfolge der Aminosäuren des Proteins. Jeweils drei nebeneinanderliegende Nukleotide bilden ein Codon, mit dessen Hilfe sich eine spezifische Aminosäure, die in ein Protein eingebaut werden soll, eindeutig bestimmen lässt.
1.1. Genexpression
Die Expression eines Gens beschreibt dessen Aktivität zur Bildung des von dem Gen kodierten Genprodukts (insbesondere Proteine oder RNA-Moleküle).
Die Expression von Genen ist nicht im gesamten Körper gleich. Die Expression in Blut und Gehirn korreliert nur mäßig (Pearson’s r von 0,24-0,64), wobei 35 bis 80 % der Gene in beiden Geweben exprimiert werden.1 Dennoch scheinen ca. 90 % der gewichteten Gen-Gen-Interaktionsnetzwerke des PFC-Transkriptoms auch im peripheren Blut erhalten zu bleiben. Gehirn und Blut weisen zudem eine signifikante Überlappung in Bezug auf exprimierte quantitative Merkmalsloki auf, was darauf hindeutet, dass gemeinsame genetische Effekte (wenn auch mit geringer Effektgröße) die Vergleichbarkeit der Genexpression in Blut und Gehirn teilweise erklären können. Auf dieser Basis versuchen Computermodelle anhand der Blut-Genexpression die Genexpression in Geweben zu berechnen.2
1.2. SNP: Einzelnukleotid-Polymorphismen
Einzelnukleotid-Polymorphismen (SNP, ausgesprochen „Snips“) sind die häufigste Form genetischer Variationen beim Menschen. Ein SNP ist eine DNA-Abweichung in einem Nukleotid. Beispiel: an einer Stelle der DNA wird das Nukleotid Cytosin (C) durch das Nukleotid Thymin (T) ersetzt.3
SNP finden sich in der gesamten menschlichen DNA an rund jeder 1.000sten Stelle. Damit finden sich im menschlichen Genom rund 4 bis 5 Millionen SNPs.
Bislang wurden weltweit mehr als 600 Millionen SNPs gefunden.
Damit ein SNP einen Namen bekommt (z.B. rs11420276) muss es bei mindestens 1 Prozent der Bevölkerung vorkommen.
Die meisten SNP finden sich nicht in den Genen, sondern in der DNA zwischen den Genen.
Die meisten SNPs haben keinen Einfluss auf die Gesundheit oder Entwicklung. SNPs innerhalb eines Gens oder in einer regulatorischen Region in der Nähe eines Gens können die Funktion des Gens beeinflussen und dadurch eine direktere Rolle bei Krankheiten spielen.
1.2. Alternatives Spleißen (AS)
Alternatives Spleißen (AS) ist der Prozess des Spleißens der Vorläufer-Boten-RNA (pre-mRNA) und ein wichtiger Mechanismus zur Erhöhung der Transkriptvielfalt.4 AS wird durch SNPs gesteuert, was über komplexe Mechanismen zu Variationen der Merkmale führt.5.
2. DNA (Desoxyribonukleinsäure)
Eine doppelstrangige Verkettung von 4 möglichen Nukleotiden.
Die DNA befindet sich meist im Zellkern als Chromosomen (nukleäre DNA = nDNA) und trägt bei fast allen Lebewesen die Erbinformation. Ein geringer Teil der DNA befindet sich in den Mitochondrien (Zellkraftwerke) als mitochondriale DNA (mtDNA).
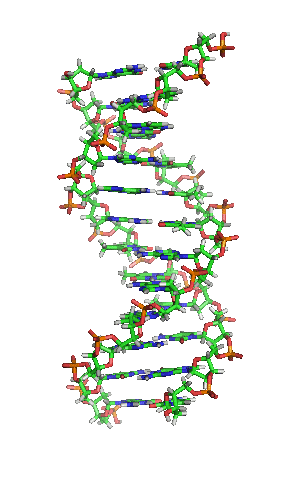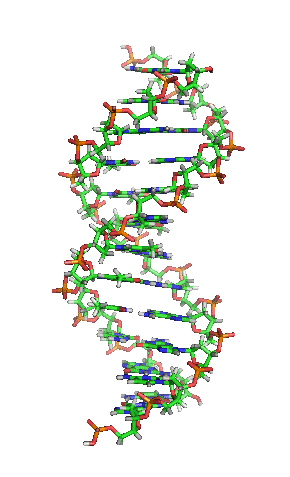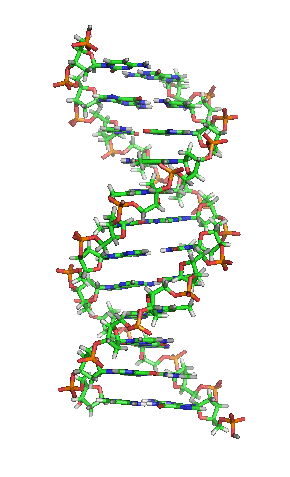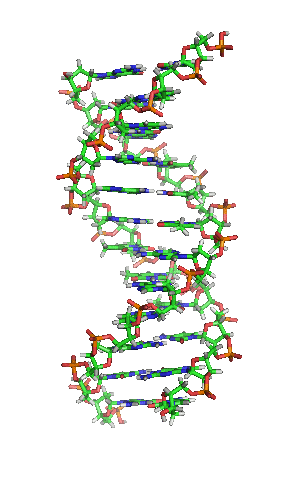
Modell einer DNA. CC BY-SA 3.0
3. RNA (Ribonukleinsäure)
Eine meist einzelstrangige Verkettung von 5 möglichen Nukleotid-Arten.
RNA ist bei bestimmten Viren der Träger der Erbinformation. Es gibt RNA, die (Erb)Informationen weitergibt (codierende RNA) und solche, die dies nicht tut.
Allein in 2017 und 2018 wurden jährlich je 2000 neue RNA-Formen entdeckt. PubMed findet in den Jahren 2014 bis 2018 zum Suchbegriff RNA rund 250.000 neu erschienene wissenschaftliche Artikel. Zum Vergleich: zum Stichwort ADHD (englisch für ADHS) waren es knapp 11.500.
3.1. Codierende RNA (mRNA)
mRNA kopiert die in einem Gen liegende Information, vermittelt sie an das Ribosom, wo mittels dieser Information die Proteinbiosynthese stattfindet.
1,2 % der RNA sind große, proteincodierende mRNA.
Formen:6
- NSD transcript
- capped mRNA
- consensus mRNA
- edited mRNA
- exemplar mRNA
- mRNA with frameshift
- monocistronic mRNA
- polyadenylated mRNA
- polycistronic mRNA
- recoded mRNA
- trans spliced mRNA
3.2. Nicht-codierende RNA (ncRNA)
Es gibt viele Arten nichtcodierender RNA.
ncRNAs werden von der DNA transkribiert, aber nicht in Proteine übersetzt. Sie beeinflussen die Zellfunktionen und die Pathogenese von Krankheiten mittels Regulierung der Genexpression durch Transkription und Translation.7, z.B. durch8
- Chromatinumbau
- Histonmodifikation
- Bindung von Transkriptionsfaktoren
- mRNA-Abbau
- mRNA-Translationshemmung
- Interaktion mit Spleißfaktoren
- Interaktion mit RNA-bindenden Proteinen
Als kompetitive endogene RNAs (ceRNAs) sequestrieren sie miRNAs und modulieren die Expression von mRNAs.7
Formen nichtcodierender RNA:910
- Housekeeping ncRNA8
- werden in allen Zellen in großen Mengen exprimiert
- sind an allgemeinen zellulären Prozessen wie der Proteinsynthese, dem RNA-Spleißen und der RNA-Modifikation beteiligt
- ribosomale RNAs (rRNAs)
- Transfer-RNAs (tRNAs)
- kleine nukleare RNAs (snRNAs)
- kleine nukleolare RNAs (snoRNAs)
- TERC (Telomerase-RNAs)
- tRNA-Derived Fragments (tRFs)
- tRNA-Hälften (tiRNAs), während
- regulatorische ncRNAs8
- regulieren gewebespezifisch die Genexpression
- Mikro-RNAs (miRNAs)
- kleine interferierende RNAs (siRNAs)
- PI-WI-interagierende RNAs (piRNAs)
- Enhancer-RNAs (eRNAs)
- zirkuläre RNAs (circRNAs)
- lange nicht-kodierende RNAs (lncRNAs)
- Y-RNAs (YRNAs)
- asRNA (Antisense-RNA)
- reguliert die Genexpression
- wird aus dem kodierenden DNA-Strang und nicht aus dem Matrizen-DNA-Strang transkribiert. Ist daher komplementär zu mRNA.
- circRNA (zirkuläre RNA)7
- bindet an miRNA. Ist dadurch an der Regulation zellulärer Prozesse beteiligt, wie z.B. Proliferation oder Apoptose (Zelltod)
- Regulierung der Genexpression, Sponge-miRNAs, Modulation der neuronalen Entwicklung, synaptische Plastizität 11
- class I RNA
- class II RNA
- enhancerRNA
- könnte regulatorische Funktion haben
- guide RNA
- hnRNA (heterogene Kern-RNA, heterogeneous nuclear RNA)/ prä-mRNA (pre-mRNA, precursor mRNA)
- Vorstufe der reifen mRNA
- lncRNA (long non-coding RNA)
- länger als 200 Nukleotide
- Gerüst-DNA-Chromatin-Komplexe, Inaktivierung des X-Chromosoms, Telomerregulation, Imprinting11
- long noncoding RNA7
- miRNAs (microRNAs)
- es sind rund 2.600 verschiedene miRNA beim Menschen bekannt8
- eng verwandt mit siRNAs
- regulieren zelluläre Prozesse wie z.B. Proliferation und Zelltod mit
- miRNAs fungieren als posttranskriptionelle Gen-Silencer, indem sie mit Ziel-mRNAs interferieren und ihre Aktivität hemmen1213 MicroRNAs (miRNAs) regulieren dadurch in der Regel die Genexpression herunter.
- Die Interaktion zwischen miRNAs und Messenger-RNAs (mRNAs) führt zum Abbau oder zur Unterdrückung der Translation der Ziel-mRNA. Dies macht sie zu einem wesentlichen Faktor des posttranskriptionellen Regulationsmechanismus11
- Bei der RNA-Interferenz (RNAi) blockiert die Einführung doppelsträngiger RNA (dsRNA) die Genexpression
- piRNA (Piwi-interacting RNA)
- 26–31 Nukleotide lang
- bilden Komplexe mit PIWI-Proteinen, die am epigenetischen und posttranskriptionellen Silencing in Keimzellen beteiligt sind
- Transposon-Repression, DNA-Methylierung11
- Riboswitches
- dienen der Genregulation
- aktivierend oder reprimierend
- dienen der Genregulation
- Ribozyme
- katalytisch aktive RNA-Moleküle
- katalysieren chemische Reaktionen, so wie Enzyme
- rRNA (ribosomale RNA)
- trägt keine genetische Information (wie tRNA)
- ist am Aufbau des Ribosoms beteiligt
- ist bei Knüpfung der Peptidbindung auch katalytisch aktiv
- rRNA cleavage RNA
- rasiRNA (repeat associated small interfering RNA)
- RNase MRP RNA
- essentiell für die katalytische Aktivität von RNase MRP.
RNase MRP ist ein enzymatisch aktives Ribonukleoprotein, das in den Mitochondrien bei der Initiierung der mitochondrialen DNA-Replikation mitwirkt und im Zellkern an der Vorläufer-rRNA-Prozessierung beteiligt ist, wo es den internen transkribierten Spacer 1 zwischen 18S- und 5,8S-rRNAs spleißt.
- essentiell für die katalytische Aktivität von RNase MRP.
- RNase P RNA
- scRNA (small cytoplasmic RNA)
- scaRNA (small cajal body-specific RNA)
- finden sich in den Cajal Bodies
- Unterklasse der snoRNA
- steuern die Modifikation (Methylierung und Pseudouridylierung) der RNA-Polymerase-II-transkribierten spliceosomalen RNAs U1, U2, U4, U5 und U12.
- scaRNA1 (small cajal body-specific RNA 1, ACA35)
- scheint an der Pseudouridylierung von U2-spliceosomaler RNA am Rest U89 beteiligt zu sein
- shRNA
- siRNA (small interfering RNA)
- entsteht bei einem als als RNAi (RNA Interference) bezeichneten Signalweg der Zelle
Dabei wird dsRNA (doppelsträngige RNA, double-stranded RNA) durch das Enzym Dicer in viele kleine siRNAs aufgeteilt und in RISC (RNA-induced silencing complex, ein Enzymkomplex) eingebaut. Mittels der enthaltenen RNA-Fragmente bindet RISC an DNA (z.B. an Genbereiche) oder an mRNA und kann diese damit „abschalten“. - siRNAs sind an verschiedenen Zellvorgängen und Krankheiten beteiligt.
- entsteht bei einem als als RNAi (RNA Interference) bezeichneten Signalweg der Zelle
- small RNA7
- snRNA (small nuclear-RNA)
- finden sich im Zellkern
- verantwortlich für das Spleißen der hnRNA am Spleißosom.
- snoRNA (small nucleolar-RNA)
- finden sich im Nukleolus
- eng verwandt mit scaRNAs
- small regulatory ncRNA
- SRP RNA
- snRNA (small nuclear-RNA)
- tRNA (Transfer-RNA)
- codiert keine genetische Information
- Hilfsmolekül bei der Proteinbiosynthese
- nimmt einzelne Aminosäure aus dem Cytoplasma auf und transportiert sie zum Ribosom
- tRNA wird durch ein bestimmtes RNA-Gen codiert
- tasiRNA
- telomerase RNA
- telomeric transcript
- three prime overlapping ncRNA
- tsRNA
- Regulierung von Genexpression, Zellprozesse, Stressreaktionen und Immunsystem11
- TSSa RNA
- Sicherung der Transkription (?)11
- vault RNA
- Y RNA
4. Nukleotide
Nukleotide bestehen jeweils aus einer Base, einem Zucker und einem Phosphat:
- Base (fünf mögliche Basen)
- Adenin (A)
- Guanin (G)
- Cytosin (C)
- Thymin (T)
- Uracil (U) (in DNA stattdessen nur Thymin)
- Zucker
- Ribose (D-Ribofuranose) oder
- Desoxyribose (2-Desoxy-D-ribofuranose)
- Phosphat
- mindestens eine Phosphatgruppe
5. Proteine (Eiweiße)
Rund 1 % der menschlichen Gene codiert Proteine.
Proteine sind erforderlich für die biologische Entwicklung eines Lebewesens und den Zellstoffwechsel.
Menschliche Proteine können aus 21 verschiedenen α-Aminosäuren entstehen. Die α-Aminosäuren verketten sich untereinander durch Peptidbindungen zu einem Polymer (Polypeptide). Diese Polypeptide falten sich im wässrigen Milieu zum nativen Protein auf.
Die Biosynthese von Proteinen findet in allen Zellen an den Ribosomen anhand von mRNA statt, die als genetische Information den Bauplan von Proteinen übermitteln.
Die Abfolge der Basen der mRNA codiert in Tripletts (Codons) die Sequenz der (proteinogenen) Aminosäure. Nach der Translation können die Seitenketten von manchen im Protein eingebauten Aminosäuren noch verändert werden.
Eine Studie fand eine Korrelation von ADHS mit den Proteinen Lysosomal Pro-X carboxypeptidase und Alpha-2-antiplasmin im Blutplasma.14
6. Aminosäuren (Aminocarbonsäuren)
Aminosäuren sind chemische Verbindungen mit einer Aminogruppe (enthält Stickstoff) und einer Carbonsäuregruppe (enthält Kohlenstoff), die in allen Lebewesen vorkommen.
Aminosäuren sind die Bausteine von Proteinen (Eiweiß).
Sie dienen primär dem Aufbau von Körpergewebe und sind die Endstufen des Abbaus von Eiweiß.
Essentielle Aminosäuren können von einem Organismus nicht selbst hergestellt werden und müssen deshalb über die Nahrung aufgenommen werden.
Aminosäuren lassen sich nach verschiedenen Kategorien unterscheiden:
6.1. Arten von Aminosäuren
6.1.1. nach Aufbau
- Aminosäuren nach Aufbau
- α-Aminosäuren (2-Aminocarbonsäuren, z.B. Glycin)
- β-Aminosäuren (3-Aminocarbonsäuren, z.B. β-Alanin)
- γ-Aminosäuren (4-Aminocarbonsäuren, z.B. γ-Aminobuttersäure = GABA)
6.1.2. Aminosäuren nach Funktion
- Proteinogenität
Proteinogenität bedeutet, dass die Aminosäure als Baustein von Proteinen dient- Proteinogene Aminosäuren
- α-Aminosäuren, die Bausteine der Proteine sind, beim Menschen 21 verschiedene
Name / Häufigkeit in Proteinen / essentiell:- Alanin (9,0 %)
- Arginin (4,7 %) teilessentiell
- Asparagin (4,4 %)
- Asparaginsäure (5,5 %)
- Cystein (2,8 %) für Kinder und Schwangere essentiell
- Glutamin (3,9 %)
- Glutaminsäure (6,2 %)
- Glycin (7,5 %)
- Histidin (2,1 %) teilessentiell
- Isoleucin (4,6 %) essentiell
- Leucin (7,5 %) essentiell
- Lysin (7,0 %) essentiell
- Methionin (1,7 %) essentiell
- Phenylalanin (3,5 %) essentiell
- Prolin (4,6 %)
- Pyrrolysin (nichtkanonisch, nur bei Bakterien)
- Selenocystein (nichtkanonisch)
- Serin (7,1 %)
- Threonin (6,0 %) essentiell
- Tryptophan (1,1 %) essentiell
- Tyrosin (3,5 %) für Kinder und Schwangere essentiell
- unbekannte Aminosäure (unklar, ob Aminosäure)
- Valin (6,9 %) essentiell
- α-Aminosäuren, die Bausteine der Proteine sind, beim Menschen 21 verschiedene
- nichtproteinogene Aminosäuren
- natürlich vorkommende (= biogene / organogene) Aminosäuren
- mehr als 400 nichtproteinogene Aminosäuren mit biologischen Funktionen
- D-Aminosäuren (selten) sind spezielle Untergruppe
- synthetisch erzeugte / theoretisch mögliche Aminosäuren
- erheblich größere Anzahl
- natürlich vorkommende (= biogene / organogene) Aminosäuren
- Proteinogene Aminosäuren
- Neurotransmitter
- einige Aminosäuren fungieren als Neurotransmitter
- GABA
- Glycin
- ebenso einige Abbauprodukte von Aminosäuren
- einige Aminosäuren fungieren als Neurotransmitter
- Hormone
- Gewebsmediatoren
6.1.3. Aminosäuren nach Entstehung
- natürlich vorkommend (= biogen / organogen)
- synthetisch erzeugt
Tylee DS, Kawaguchi DM, Glatt SJ. On the outside, looking in: a review and evaluation of the comparability of blood and brain “-omes”. Am J Med Genet B Neuropsychiatr Genet. 2013 Oct;162B(7):595-603. doi: 10.1002/ajmg.b.32150. PMID: 24132893. REVIEW ↥
Hess JL, Quinn TP, Zhang C, Hearn GC, Chen S; Neuropsychiatric Consortium for Analysis and Sharing of Transcriptomes; Kong SW, Cairns M, Tsuang MT, Faraone SV, Glatt SJ. BrainGENIE: The Brain Gene Expression and Network Imputation Engine. Transl Psychiatry. 2023 Mar 22;13(1):98. doi: 10.1038/s41398-023-02390-w. PMID: 36949060; PMCID: PMC10033657. ↥
medlineplus.gov: What are single nucleotide polymorphisms (SNPs)? ↥
Wang J, Zhu QW, Mai JH, Zhang S, Wang Y, Liang J, Zhou JY (2024): A multi-omics study of brain tissue transcription and DNA methylation revealing the genetic pathogenesis of ADHD. Brief Bioinform. 2024 Sep 23;25(6):bbae502. doi: 10.1093/bib/bbae502. PMID: 39406522; PMCID: PMC11479714. ↥
Qi T, Wu Y, Fang H, Zhang F, Liu S, Zeng J, Yang J (2022): Genetic control of RNA splicing and its distinct role in complex trait variation. Nat Genet. 2022 Sep;54(9):1355-1363. doi: 10.1038/s41588-022-01154-4. PMID: 35982161; PMCID: PMC9470536. ↥
Gao Y, Takenaka K, Xu SM, Cheng Y, Janitz M (2025): Recent advances in investigation of circRNA/lncRNA-miRNA-mRNA networks through RNA sequencing data analysis. Brief Funct Genomics. 2025 Jan 15;24:elaf005. doi: 10.1093/bfgp/elaf005. PMID: 40251826; PMCID: PMC12008121. REVIEW ↥ ↥ ↥ ↥ ↥
Karaivazoglou K, Triantos C, Aggeletopoulou I (2025): Non-Coding RNAs in Neurodevelopmental Disorders-From Diagnostic Biomarkers to Therapeutic Targets: A Systematic Review. Biomedicines. 2025 Jul 24;13(8):1808. doi: 10.3390/biomedicines13081808. PMID: 40868063; PMCID: PMC12384018. REVIEW ↥ ↥ ↥ ↥
Khoodoruth MAS, Khoodoruth WNC, Uroos M, Al-Abdulla M, Khan YS, Mohammad F (2024): Diagnostic and mechanistic roles of MicroRNAs in neurodevelopmental & neurodegenerative disorders. Neurobiol Dis. 2024 Nov;202:106717. doi: 10.1016/j.nbd.2024.106717. PMID: 39461569. REVIEW ↥ ↥ ↥ ↥ ↥ ↥
Guo H, Ingolia NT, Weissman JS, Bartel DP (2010): Mammalian microRNAs predominantly act to decrease target mRNA levels. Nature. 2010 Aug 12;466(7308):835-40. doi: 10.1038/nature09267. PMID: 20703300; PMCID: PMC2990499. ↥
Bartel DP (2009): MicroRNAs: target recognition and regulatory functions. Cell. 2009 Jan 23;136(2):215-33. doi: 10.1016/j.cell.2009.01.002. PMID: 19167326; PMCID: PMC3794896. REVIEW ↥
Cheng, Guan, Ma, Zhang, Cheng, Qi, Liang, Li, Kafle, Wen, Zhang (2020): An atlas of genetic correlations between psychiatric disorders and human blood plasma proteome. Eur Psychiatry. 2020 Feb 20;63(1):e17. doi: 10.1192/j.eurpsy.2019.6. PMID: 32093803; PMCID: PMC7315878. ↥